こんにちは、のっくんです。
今日は深層学習フレームワークPyTorchで転移学習のやり方をご紹介します。
使用するデータセットは、マラリアの細胞データを使います。
細胞データには2パターンあり、感染しているものと、感染していないものがデータセットに含まれています。
このデータはkaggleに公開されているものですので、誰でも入手可能です。
データセットの詳しい内容は以下の記事に記載してあります。
[toc]転移学習って何?
そもそも転移学習って何かと言うと、すでに学習済みのモデルとパラメータを使って深層学習する方法です。
普通の学習では、学習することで重みなどのパラメータが調整されていきますが、転移学習では事前に調整されたパラメータをそのまま(凍結させて)使います。
事前に調整されたモデルやパラメータをそのまま使用することを、転移学習と呼ぶわけですね。
データの読み込み
データの読み込みには、ImageFolderを使うと便利です。
ImageFolderを使用するためにディレクトリ構成を整える必要があります。
以下のように訓練用と検証用にフォルダを分けて、その中に感染している細胞、感染していない細胞のフォルダを作成する感じです。
train - Parasitised
- Uninfected
val - Parasitised
- Uninfected
元のデータでは2万枚ほどありましたが、その中から訓練用に1000枚、テスト用に500枚をコピーします。
コピーする際にはリスト内包表記を使って、ファイル名をリストに格納しました。
train_para_list = [os.path.join(img_path_para,i) for i in os.listdir(img_path_para)[:500]] val_para_list = [os.path.join(img_path_para,i) for i in os.listdir(img_path_para)[500:750]] train_unin_list = [os.path.join(img_path_unin,i) for i in os.listdir(img_path_unin)[:500]] val_unin_list = [os.path.join(img_path_unin,i) for i in os.listdir(img_path_unin)[500:750]]
データ拡張するために、transformsというパッケージを使用します
#画像の前処理を定義
data_transforms = {
'train': transforms.Compose([
transforms.RandomResizedCrop(224),
transforms.RandomHorizontalFlip(),
transforms.ToTensor(),
transforms.Normalize([0.485, 0.456, 0.406], [0.229, 0.224, 0.225])
]),
'val': transforms.Compose([
transforms.Resize(256),
transforms.CenterCrop(224),
transforms.ToTensor(),
transforms.Normalize([0.485, 0.456, 0.406], [0.229, 0.224, 0.225])
]),
}
#画像とラベルを読み込む
data_dir = '.'
image_datasets = {x: datasets.ImageFolder(os.path.join(data_dir, x),
data_transforms[x])
for x in ['train', 'val']}
訓練画像の場合には、
- RandomResizedCrop, ランダムな位置をランダムなサイズで切り取って、指定したサイズにリサイズします。
- RandomHorizontalFlip,50%の確率で水平方向反転します。
- Normalize, 正規化します。
テスト画像の場合には、
- Resize, 指定したサイズにリサイズします。
- CenterCrop, 指定したサイズ分中心を切り抜きます。
- Normalize, 正規化します。
上記コードではカレントディレクトリにtrainとvalというフォルダがあることが前提で、ImageFolderを使って画像を読み込みます。
転移学習
PyTorchでは、Alexnet、VGG、ResNet、SqueezeNet、Inception v3などの代表的なネットワークが使えます。
ここではAlexnetを使って転移学習をしてみます。pretrained=Trueにすることで学習済みの重みを使用します。
#ネットワークalexnetの定義 net = models.alexnet(pretrained=True) net = net.to(device) net
AlexNet(
(features): Sequential(
省略
)
(avgpool): AdaptiveAvgPool2d(output_size=(6, 6))
(classifier): Sequential(
(0): Dropout(p=0.5)
(1): Linear(in_features=9216, out_features=4096, bias=True)
(2): ReLU(inplace)
(3): Dropout(p=0.5)
(4): Linear(in_features=4096, out_features=4096, bias=True)
(5): ReLU(inplace)
(6): Linear(in_features=4096, out_features=1000, bias=True)
)
)
classifierの6番目(最終層)を見ると、out_featuresが1000になっています。
これは最終的に1000個のクラスに分類するように学習されているということです。
今回は2クラス分類なのでこれを2に変更し、最終層以外のパラメータを凍結させます。
#ネットワークのパラメータを凍結
for param in net.parameters():
param.requires_grad = False
net = net.to(device)
#最終層を2クラス用に変更
num_ftrs = net.classifier[6].in_features
net.classifier[6] = nn.Linear(num_ftrs, 2).to(device)
個人的な感想ですが、kerasよりもPyTorchの方がこの辺りのパラメータの変更が直感的で分かりやすいと思います。
これだけで転移学習の準備が完了です。
学習
細かい部分は省略しますが、学習時に学習率を変更するようにlr_schedulerを使用します。
#最適化関数
criterion = nn.CrossEntropyLoss()
optimizer = optim.SGD(net.parameters(), lr=0.01, momentum=0.9, weight_decay=5e-4)
lr_scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=10, gamma=0.1)
num_epochs = 15
for epoch in range(num_epochs):
#train
net.train()
for i, (images, labels) in enumerate(train_loader):
#省略
#val
net.eval()
with torch.no_grad():
for images, labels in test_loader:
#省略
#学習率調整
lr_scheduler.step()
学習結果をプロットしてみました。
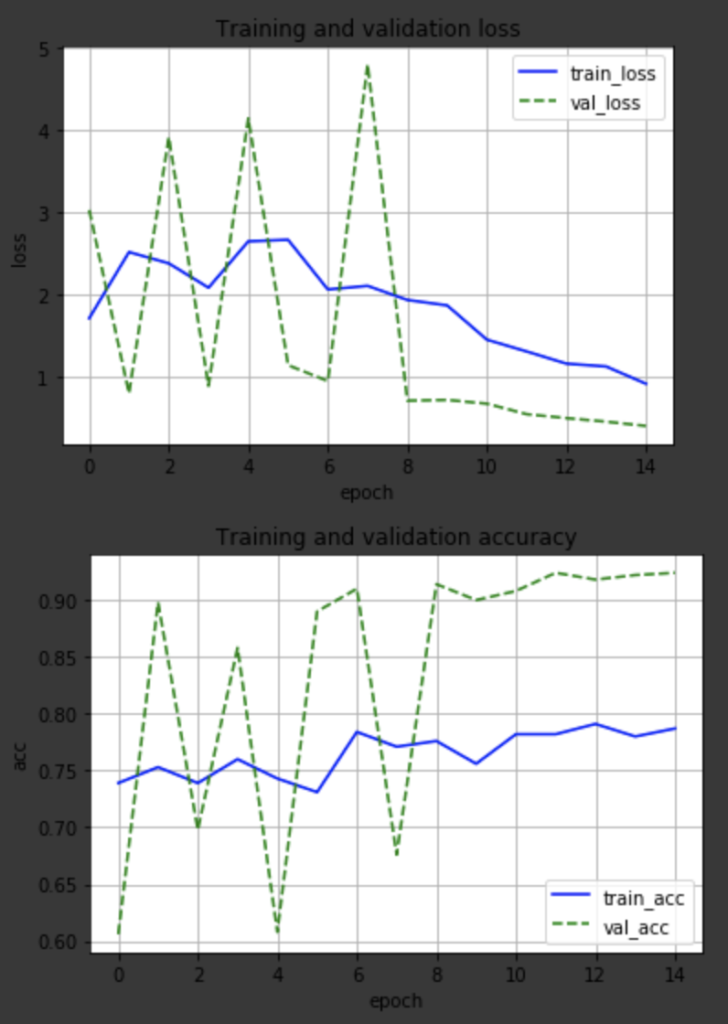
検証精度を見ると、最初の方は不安定ですが最後の方は安定して9割を超えました。