こんにちは、のっくんです。
皆さんは、マラリアってご存知ですかね。
今日の記事では、ディープラーニングを使って細胞の画像からマラリアに感染しているかを判別してみたいと思います。
[toc]
データセット
マラリアとは?
マラリアは世界100か国以上でみられる感染症で、WHOの推計では毎年3~5億人が感染、数百万人が死亡しているとされます。主な流行地はサハラ以南アフリカ、東南・南アジア、中南米、パプアニューギニア、ソロモン諸島など熱帯・亜熱帯地域です。近年では、毎年50例前後の輸入症例が報告されています。
マラリア原虫保有雌ハマダラカによる吸血時に原虫が体内に侵入し、感染が成立します。原虫の種類により三日熱、四日熱、卵形、熱帯熱に分類されます。国内にも三日熱マラリアを媒介するシナハマダラカは広く生息しています。重症型の熱帯熱マラリアを媒介するコガタハマダラカは沖縄の宮古・八重山諸島にのみ生息していますが、今後温暖化が進行すれば沖縄本島から九州南部へと生息地域が拡がる可能性もあります。
https://www.forth.go.jp/keneki/kanku/disease/dis07_03mal.html
まぁ要はハマダラカとかいう蚊に刺されると、感染して下手すると死ぬらしいです。そして日本でも沖縄にいるそうです。怖いですね。。
んで、以下のサイトにマラリアのセル(細胞)データセットがアップされています。ログインするとダウンロードできます。
https://www.kaggle.com/iarunava/cell-images-for-detecting-malaria/version/1
NIHっていう機関から取得したらしいですがNIHのサイトからダウンロードすると重いので、kaggleからダウンロードした方が良いらしい。
なにせ、27558枚もあるからね。
OpenCVで64*64のサイズにリサイズして読み込みます。
感染したパターンと感染していないパターンの2つがあります。
- 感染した細胞
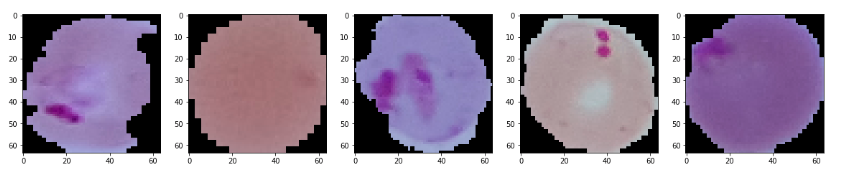
- 感染していない細胞
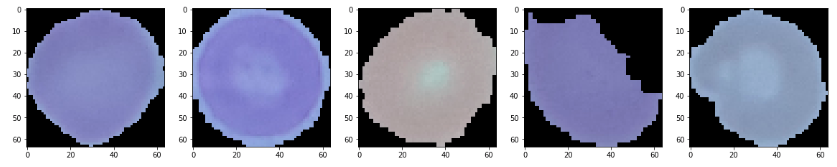
使用するCNN
使用する畳み込みニューラルネット(CNN)は以下の通り。
- 畳み込み層
畳み込み層は2つ。出力は両方とも32、カーネルサイズは3*3にします。入力サイズは、画像の縦横のサイズ、最後の3はチャンネル数で、カラーなので3です。
- プーリング
プーリングの行列のサイズを2*2に設定。入力が62*62のサイズだとしたら、この層を通過すると半分の31*31のサイズになります。
チャンネル数が最後にあるので、data_formatはチャネルラストになります。
- バッチ正規化
チャネルラストの時は、-1を指定します。
- ドロップアウト
過学習防止のためです。20%の割合で値を0にします。
- 平坦化
全結合層に行く前に、三次元配列を一次元に変換します。
- 全結合層(Dense)
全結合層の数は3つ。それぞれ活性化関数に、最初の2つはrelu、最後はsigmoidを指定しています。
ノードの数は512→256→2のように減っていきます。
model = Sequential() model.add(Convolution2D(32, (3, 3), input_shape = (SIZE, SIZE, 3), activation = 'relu')) model.add(MaxPooling2D(pool_size = (2, 2), data_format="channels_last")) model.add(BatchNormalization(axis = -1)) model.add(Dropout(0.2)) model.add(Convolution2D(32, (3, 3), activation = 'relu')) model.add(MaxPooling2D(pool_size = (2, 2), data_format="channels_last")) model.add(BatchNormalization(axis = -1)) model.add(Dropout(0.2)) model.add(Flatten()) model.add(Dense(activation = 'relu', units=512)) model.add(BatchNormalization(axis = -1)) model.add(Dropout(0.2)) model.add(Dense(activation = 'relu', units=256)) model.add(BatchNormalization(axis = -1)) model.add(Dropout(0.2)) model.add(Dense(activation = 'sigmoid', units=2)) model.compile(optimizer = 'adam', loss = 'categorical_crossentropy', metrics = ['accuracy']) model.summary()
学習
エポックは50,バッチサイズは64で動かします。結構重いので、GPUがあると良いです。
history = model.fit(np.array(X_train),
y_train,
batch_size = 64,
verbose = 2,
epochs = 50,
validation_split = 0.1,
shuffle = False)
学習後テストしてみると、そこそこの精度で見分けられました。
Test_Accuracy: 94.99%
データ拡張
ちなみにデータ拡張をすると少し精度が上がりました。
from keras.preprocessing.image import ImageDataGenerator
train_generator = ImageDataGenerator(rescale = 1/255,
zoom_range = 0.3,
horizontal_flip = True,
rotation_range = 30)
test_generator = ImageDataGenerator(rescale = 1/255)
train_generator = train_generator.flow(np.array(X_train),
y_train,
batch_size = 64,
shuffle = False)
test_generator = test_generator.flow(np.array(X_test),
y_test,
batch_size = 64,
shuffle = False)
Test_Accuracy(after augmentation): 95.41%
こんな感じ!
医者の代わりにAIが診断してくれる日が来るかも?
おわり。
参考
towardsdatascience.com,
kaggle.com,
https://www.kaggle.com/iarunava/cell-images-for-detecting-malaria/version/1
forth.go.jp,
https://www.forth.go.jp/keneki/kanku/disease/dis07_03mal.html











