この記事ではPythonの機械学習フレームワークXGBoostを使って、糖尿病の患者を推定するコードを書いてみたいと思います。
機械学習というと、K-NN(K- Nearest Neighbor)やランダムフォレスト、SVM(Support Vector Machine) が有名です。
実は私も、XGBoostを使ったことがありませんでしたが、ふとネットで見かけて使ってみたら思ったよりも精度が良かったのでご紹介したいと思います。
[toc]事前準備
まずはデータセットの収集から。
糖尿病データは以下からダウンロードしました。
https://www.kaggle.com/uciml/pima-indians-diabetes-database
次に、XGBoostをインストールします。
XGBoostは調べてみたら、conda-forgeのチャンネルにあるみたい。
いつもなら下記のようにコマンドでパッケージをインストールしています。
「conda install -c チャンネル名 パッケージ名」
でも、よくよく調べてみたらAnaconda Navigatorでも、チャンネルを追加することができたんですよ。(今更感)
channelで、以下のように”conda-forge”を入力して追加するだけです。
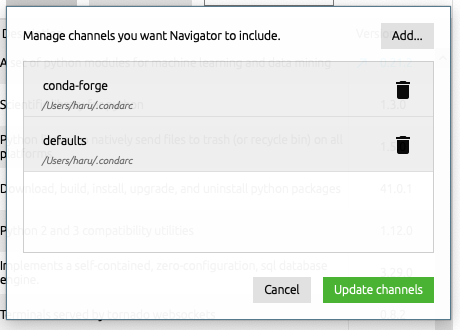
これで、Anaconda NavigatorからもXGBoostが検索&インストールできました。
学習&ハイパーパラメータ探索
データを読み込んで、特徴量とラベルに分割します。
その後にGridSearchCVを使って、ハイパーパラメータ、つまり学習時の最良なパラメータを探索します。
探索する時の可変パラメータを、variable_params, 固定パラメータを、static_paramsに格納します。
最後に、精度が最も良かった時の精度とパラメータを出力して終了です。
import numpy as np
import pandas as pd
from xgboost.sklearn import XGBClassifier
from sklearn.model_selection import GridSearchCV
data=pd.read_csv("diabetes.csv")
print(data.describe())
print(data.keys())
X_data=data.drop(["Outcome"],axis=1)
y_data=data["Outcome"]
variable_params = {'max_depth':[2,4,6,10], 'n_estimators':[5, 10, 20, 25], 'learning_rate':np.linspace(1e-16, 1 , 3)}
static_params = {'objective':'multi:softmax','num_class':4, 'silent':1}
bst_grid = GridSearchCV (
estimator = XGBClassifier(**static_params),
param_grid = variable_params,
scoring = "accuracy"
)
bst_grid.fit(X_data, y_data)
print("Best Accuracy:{}".format(bst_grid.best_score_))
for key,value in bst_grid.best_params_.items():
print("{}:{}".format(key,value))
出力は以下の通り。
Best Accuracy:0.7734375 learning_rate:0.5 max_depth:4 n_estimators:10
以下の記事で、Kerasで同じデータセットに対してディープラーニングで学習させてみたのですが、その時は73%程度でした。
より短いコードで高い精度が出たので、XGBoostさん優秀ですね。
特徴量エンジニアリング
次に、どの特徴量が重要か判別するためのコードを書いていきたいと思います。
これを特徴量エンジニアリングや分析といったりします。
DMatrixというのは、XGBoostで使うためのデータマトリックスのこと。
メモリの使用量や学習スピードが最適化されているようです。
https://xgboost.readthedocs.io/en/latest/python/python_api.html#xgboost.DMatrix
import xgboost as xgb
from sklearn.model_selection import train_test_split
import pandas as pd
%matplotlib inline
data = pd.read_csv("diabetes.csv")
print(data.describe())
print(data.keys())
X_data=data.drop(["Outcome"],axis=1)
y_data=data["Outcome"]
dtrain = xgb.DMatrix(X_data,y_data)
params = {
'objective':'binary:logistic',
'max-depth':2,
'silent':1,
'eta':0.5
}
num_rounds = 5
bst = xgb.train(params,dtrain,num_rounds)
tdump = bst.get_dump(fmap = "./featmap.txt", with_stats = True)
for trees in tdump:
print(trees)
xgb.plot_importance(bst, importance_type = 'gain', xlabel = 'Gain')
get_dumpを使用することで、パラメータをテキストファイルに書き出すことができます。
plot_importanceで、特徴量の重要度をグラフ化して出力します。(Matplotlibが必要です。)
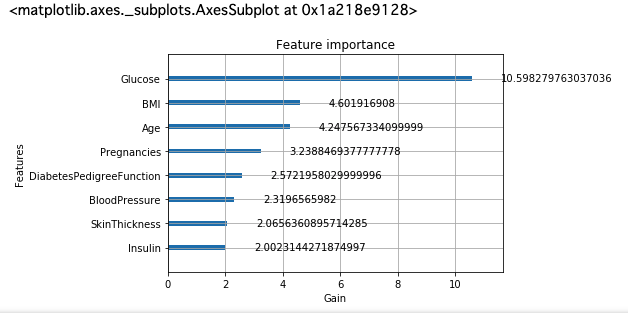
グルコース(血糖値)が糖尿病に最も関係していることがわかりました。
糖尿病の人は血糖値が高いと言われています。
血糖値だけを見てもある程度は判断できてしまうのでしょう。
BMIや年も、糖尿病と判断するのに重要だと分かりました。
このようにグラフ化してみると、何が重要な特徴量がすぐにわかって面白いですね。
おわり。
参考
https://www.kaggle.com/uciml/pima-indians-diabetes-database
https://towardsdatascience.com/model-tuning-feature-engineering-using-xgboost-ef819bccc82e